硅藻是一类重要的单细胞光合真核生物, 分布广泛, 提供了地球上约20%的初级生产力,对整个地球生物圈意义重大,三角褐指藻 (Phaeodactylum tricornutum) 是海洋硅藻的模式生物,其基因组序列于2008年公布,但目前基因组的注释仍很不完善。
蛋白基因组学 (Proteogenomics) 是利用蛋白质组学数据,尤其是高精度的串联质谱数据, 结合基因组和转录组数据对基因组进行深度注释。中国科学院水生生物研究所葛峰课题组采用蛋白基因组学的研究策略和方法,完成了模式蓝细菌的基因组深度解析 (PNAS,2014,111(52):E5633-E5642) 并开发了针对原核生物的蛋白基因组学专业分析软件GAPP(Molecular & Cellular Proteomics, 2016; 15 (11): 3529-3539)。
在上述工作基础上,葛峰课题组对真核模式硅藻三角褐指藻的基因组进行了深度解析并构建了蛋白质组精细图谱,相关成果以“Genome annotation of a model diatom Phaeodactylum tricornutum using an integrated proteogenomic pipeline”为题于近日发表在Molecular Plant杂志上。
葛峰课题组通过整合基因组、转录组、ESTs序列等多组学数据,并对数据库进行了缩减,得到去冗余的三角褐指藻蛋白基因组学数据库;通过整合基于蛋白和肽段的样品预分离、双酶切和高分辨质谱分析技术,获得高质量的质谱数据;质谱数据的鉴定整合了多种搜索引擎的结果,提高了蛋白鉴定的深度与覆盖度;并采用更为严格的肽段假阳性控制策略,从而提高鉴定结果的可信度;通过开发新的算法,实现了真核生物中新的可变剪切体的发现与点突变基因的鉴定。
本研究精准鉴定到6628个已注释的编码基因;对未鉴定到的已注释基因的深入分析发现,有1895个基因可能并不编码蛋白;发现了606个新的蛋白编码基因并校正了506个已注释的编码基因,其中有56个新发现的蛋白编码基因,在之前的研究中被错误预测为长链非编码RNA(LncRNA);鉴定到 268个可能具有重要功能的微小短肽(micropeptides),21个新的可变剪切体,并修正了73个已注释基因的可变剪切位点以及58个发生氨基酸突变的基因;通过将开放式与限定式检索相结合的策略,对三角褐指藻中的翻译后修饰进行系统鉴定,发现了20多种不同种类的蛋白质翻译后修饰,这些修饰可能参与调控细胞内众多的生物学过程并在细胞的逆境适应中起着重要作用。通过以上工作的完成,实现了三角褐指藻基因组的深度注释和蛋白质组精细图谱的构建。
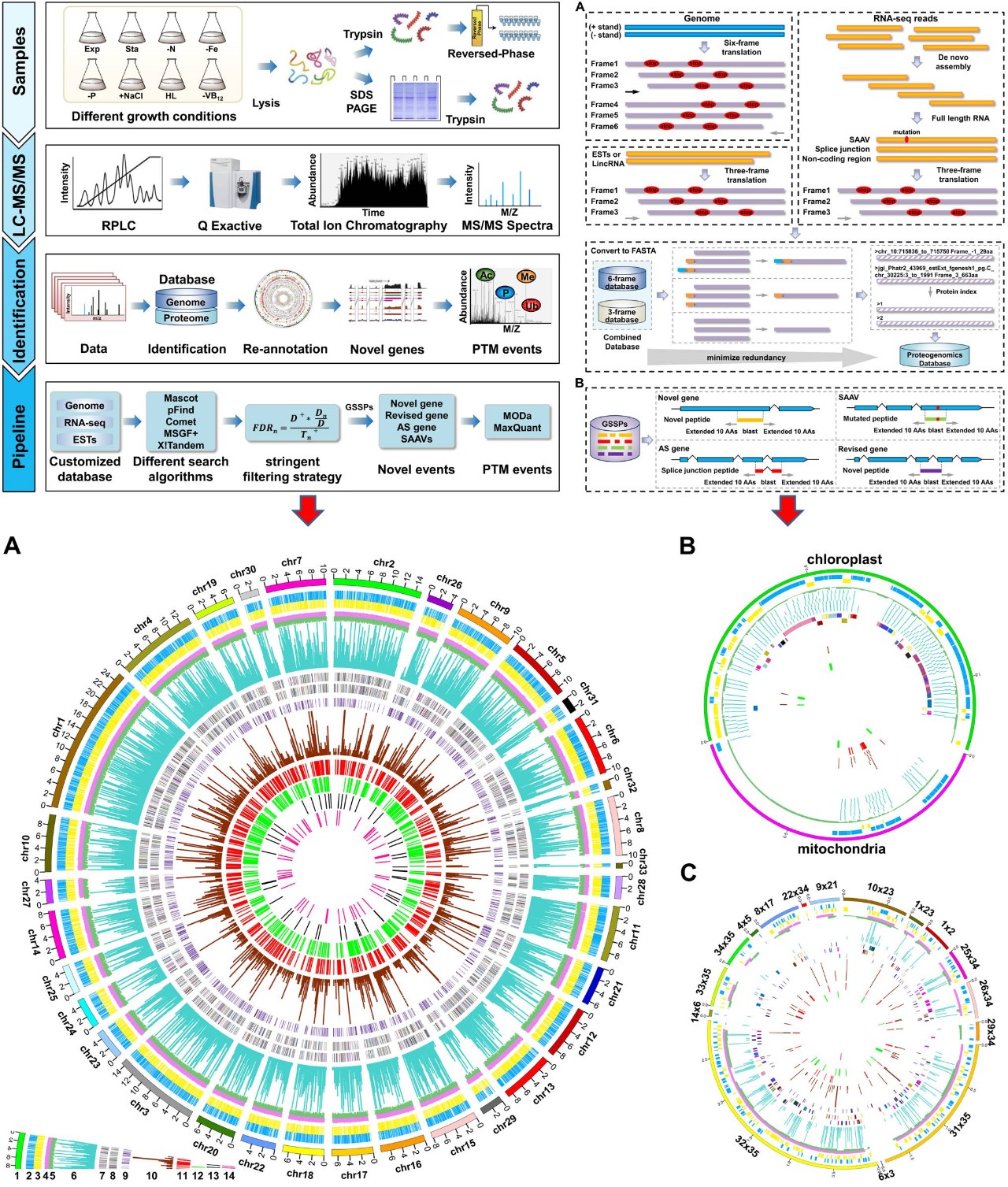
此外,在以上工作的基础上,本研究还建立了完整的构建真核模式生物的蛋白质组精细图谱的实验技术和分析流程,可适用于各种已经测序的真核生物,成为解读真核生物基因组及其功能分析的重要工具。
该论文的第一作者是水生所杨明坤高级实验师,通讯作者是葛峰研究员,该研究得到了国家重点研发计划 (2016YFA0501304)的资助。
转自:BioArt植物




